当我想查看软件运行结束后得到的BAM, BigWig, BED和GTF文件时,我都需要先把他们下载到本地,然后用IGV打开。每每这个时候,我就会非常痛苦,因为我懒得下载。
为了解决这个问题,我基于igv.js 写了一个Python脚本,可以根据提供的参考基因组,bam文件输出一个网页,然后通过利用 node.js 或者 Python的网页服务器打开一个端口,直接在网页上进行查看。
代码地址: https://github.com/xuzhougeng/myscripts/blob/master/igv_web.py
可以下面这行代码进行下载脚本, 例如我放在了家目录
1 | wget https://raw.githubusercontent.com/xuzhougeng/myscripts/master/igv_web.py |
1 | python3 ~/igv_web.py -r ref/genome.fa -m 01-read-align/*.bam -b feature.bed |
接着用python的 SimpleHTTPServer 模块启动一个网页服务器
1 | /usr/bin/python -m SimpleHTTPServer 9999 |
当然更加推荐使用npm的http-server,通过网页进行访问
1 | npx http-server ./ -p 9999 -a 0.0.0.0 |
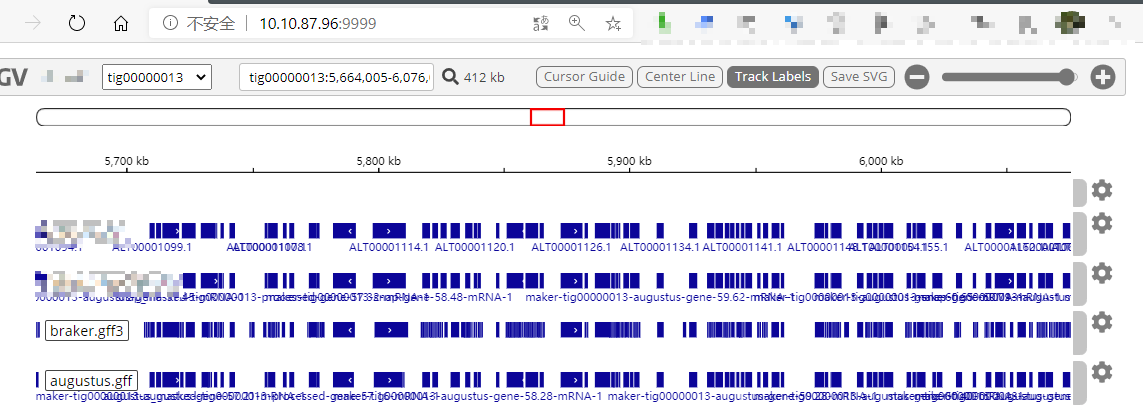
效果如下