Seurat 3.X版本能够整合scRNA-seq和scATAC-seq, 主要体现在:
- 基于scRNA-seq的聚类结果对scATAC-seq的细胞进行聚类
- scRNA-seq和scATAC-seq共嵌入(co-embed)分析
整合步骤包括如下步骤:
- 从ATAC-seq中估计RNA-seq表达水平,即从ATAC-seq reads定量基因表达活跃度
- 使用LSI学习ATAC-seq数据的内部结构
- 鉴定ATAC-seq和RNA-seq数据集的锚点
- 数据集间进行转移,包括聚类的标签,在ATAC-seq数据中推测RNA水平用于共嵌入分析
数据下载
测试数据下载地址:
scATAC-seq: h5格式
scATAC-seq metadata: csv文件
scRNA-seq: h5格式
可以复制下载链接到浏览器下载,也可以直接在R语言用download.file中进行下载。
1 | # 下载peak |
基因活跃度定量
首先,先将peak矩阵转成基因活跃度矩阵。Seurat做了一个简单的假设,基因活跃度可以通过简单的将落在基因区和其上游2kb的count相加得到基因活跃度,并且这个结果Cicero等工具返回gene-by-cell矩阵是类似的。
1 | library(Seurat) |
activity.matrix是一个dgCMatrix对象,其中行为基因,列为细胞。因此如果对于Cicero的输出结果,只要提供相应的矩阵数据结构即可。
设置对象
下一步,我们要来设置Seurat对象,将原始的peak counts保存到assay中,命名为”ATAC”
1 | pbmc.atac <- CreateSeuratObject(counts = peaks, assay = "ATAC", project = "10x_ATAC") |
增加基因活跃度矩阵,命名为”ACTIVITY”.
1 | pbmc.atac[["ACTIVITY"]] <- CreateAssayObject(counts = activity.matrix) |
增加细胞的元信息,该信息来自于scATAC-seq的CellRanger处理的singlecell.csv
1 | meta <- read.table(singlecell_file, sep = ",", header = TRUE, row.names = 1, |
过滤掉scATAC-seq数据中总count数低于5000的细胞,这个阈值需要根据具体实验设置
1 | pbmc.atac <- subset(pbmc.atac, subset = nCount_ATAC > 5000) |
数据预处理
为了找到scATAC-seq数据集和scRNA-seq数据集之间的锚定点,我们需要对基因活跃度矩阵进行预处理
设置pbmc.atac的默认Assay为”ACTIVITY”, 然后寻找高表达的基因,对基因活跃度矩阵进行标准化和Scale。
1 | DefaultAssay(pbmc.atac) <- "ACTIVITY" |
同样的,我们还需要对peak矩阵进行处理。这里我们用的隐语义(Latent semantic indexing, LSI)方法对scATAC-seq数据进行降维。该步骤学习scRNA-seq数据的内部结构,并且在转换信息时对锚点恰当权重的决定很重要。
根据 Cusanovich et al, Science 2015提出的LSI方法,他们搞了一个RunLSI函数。LSI的计算方法为TF-IDF加SVD。
我们使用在所有细胞中至少有100个read的peak,然后降维到50。该参数的选择受之前的scATAC-seq研究启发,所以没有更改,当然你你可以把它改了。
1 | DefaultAssay(pbmc.atac) <- "ATAC" |
注: 要将pbmc.atac的默认assay切换成”ATAC”, 非线性降维可以选择UMAP或者t-SNE。
我们之前使用过Seurat对scRNA-seq数据进行预处理和聚类,下载地址为Dropbox。
1 | pbmc.rna <- readRDS("pbmc_10k_v3.rds") |
将scRNA-seq和scATAC-seq共同展示,对一些骨髓(myeloid)和淋巴(lymphoid)PBMC中比较常见的聚类,其实是能从图中看出来。
1 | p1 <- DimPlot(pbmc.atac, reduction = "tsne") + |
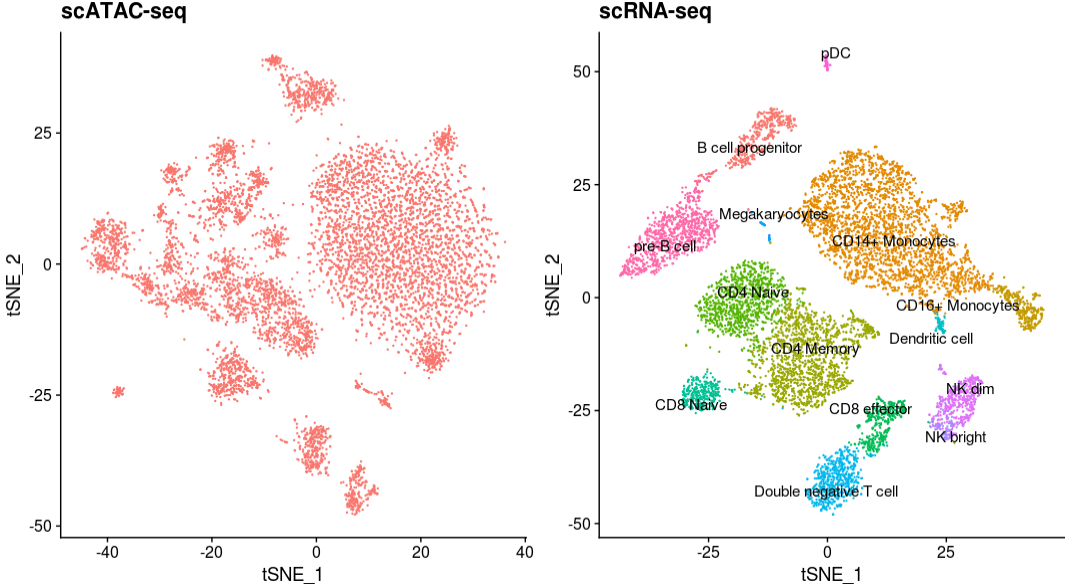
现在,我们用FindTransferAnchors鉴定scATAC-seq数据集和scRNA-seq数据集的锚点,然后根据这些锚点将 10K scRNA-seq数据中鉴定的细胞类型标记转移到scATAC-seq细胞中。
1 | transfer.anchors <- FindTransferAnchors(reference = pbmc.rna, |
Seurat比较的是scRNA-seq表达量矩阵和scATAC-seq中基因活跃度矩阵,利用CCA降维方法比较两者在scRNA-seq中的高变异基因的关系
为了转移细胞类群的编号,我们需要一组之前注释过的细胞类型,作为TransferData的 refdata 参数输入。TransferData本质上采用的是KNN算法,利用距离未知类型细胞的最近N个已知细胞所属的类型来定义该细胞。weight.reduction参数是用来选择设置权重的降维。
1 | celltype.predictions <- TransferData(anchorset = transfer.anchors, |
我们可以检查每个细胞预测得分的分布情况,选择性的过滤哪些得分比较低的细胞。我们发现超过95%的细胞得分大于或等于0.5.
1 | hist(pbmc.atac$prediction.score.max) |
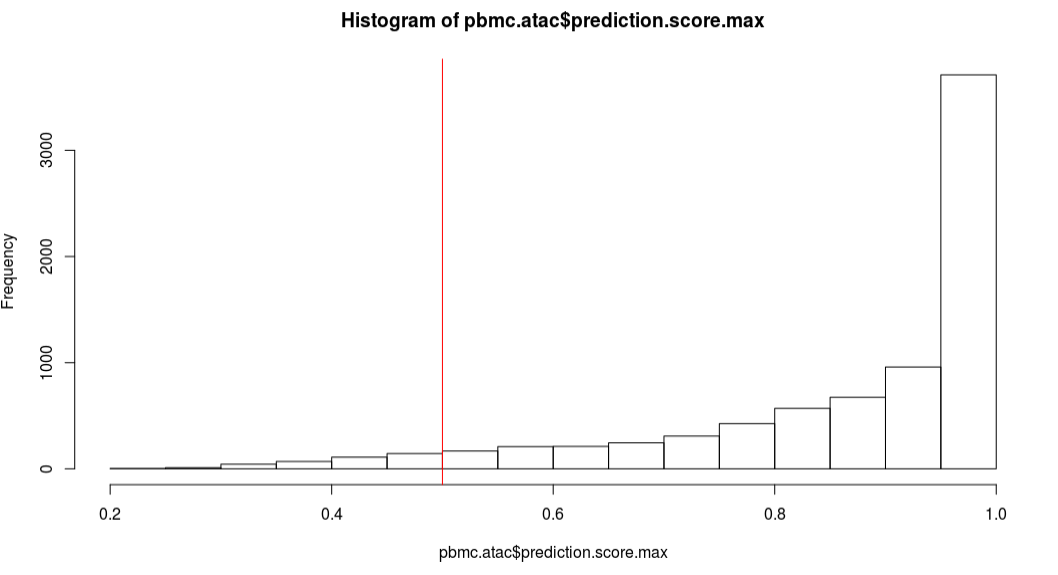
1 | table(pbmc.atac$prediction.score.max > 0.5) |
之后,我们就可以在UMAP上检查预测的细胞类型的分布,检查细胞类型在scRNA-seq和scATAC-seq中的相对位置
1 | pbmc.atac.filtered <- subset(pbmc.atac, |
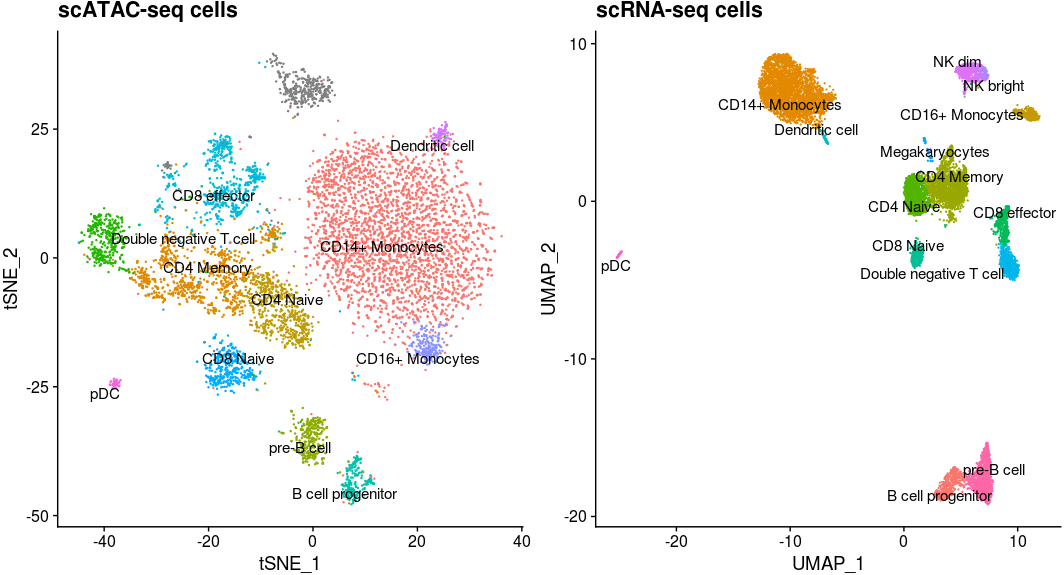
在转移细胞类型标记之后,我们就可以进行细胞特意水平上的下有分析。举个例子,我们可以去找一些某些细胞类型特异的增强子,寻找富集的motif。目前这些分析Seurat还不直接支持,还在调试中。
共嵌入(co-embedding)
最后,如果你想将所有的细胞一同展示,可以将scRNA-seq和scATAC-seq数据嵌入到相同的低维空间。
我们使用之前的锚点从scATAC-seq细胞中推断RNA-seq的值,后续分析就相当于两个单细胞数据的分析流程。
注意: 这一步只是为了可视化,其实不做也行。
选择用于估计的基因,可以是高变动基因,也可以是所有基因。
1 | # 只选择高变动的基因作为参考 |
之后利用TransferData推断scATAC-seq在这些基因中的可能值,输出结果就是ATAC细胞对应的scRNA-seq矩阵
1 | imputation <- TransferData(anchorset = transfer.anchors, |
合并两个的结果,然后就是scRNA-seq的常规分析。
1 | coembed <- merge(x = pbmc.rna, y = pbmc.atac) |
在t-SNE上绘制结果
1 | p1 <- DimPlot(coembed, reduction="tsne", group.by = "tech") |
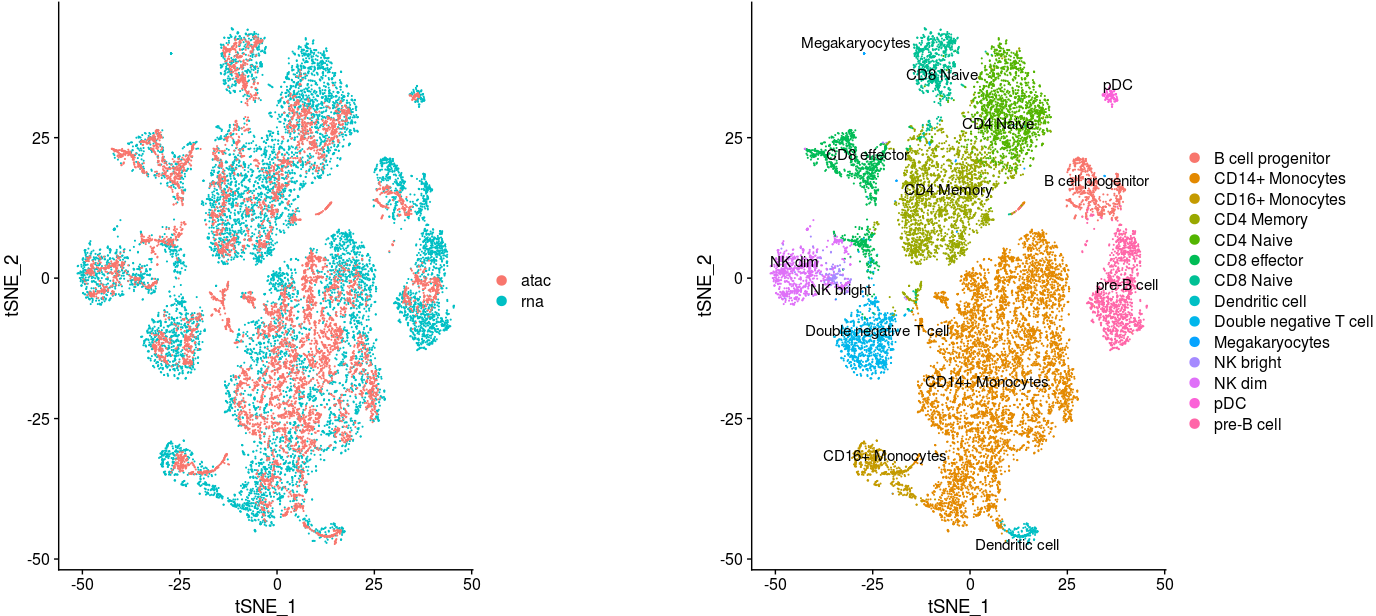
从上面的结果中,你可能会发现某些细胞只有在一类技术中存在。举个例子,从巨噬细胞(megakaryocytes)到成熟的血小板细胞(pletelet)由于没有细胞核,无法被scATAC-seq技术检测到。我们可以单独展示每个技术,进行检查
1 | DimPlot(coembed, reduction="tsne", split.by = "tech", group.by = "celltype", label = TRUE, repel = TRUE) + NoLegend() |
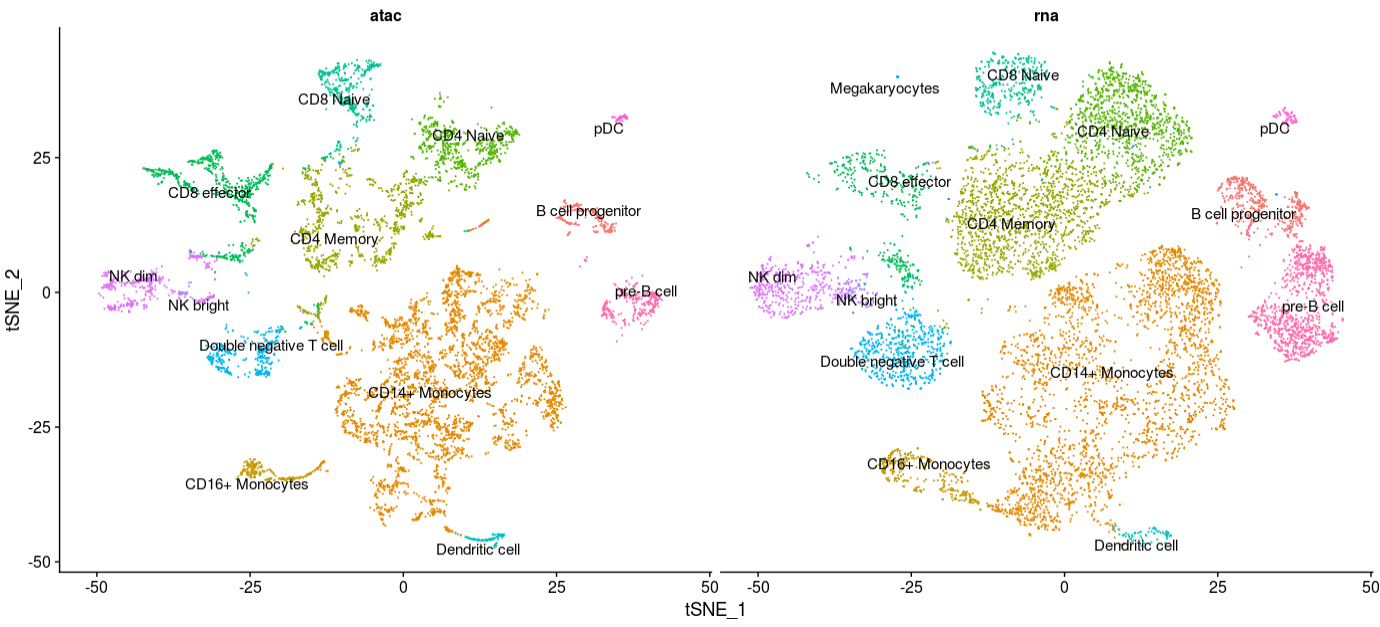
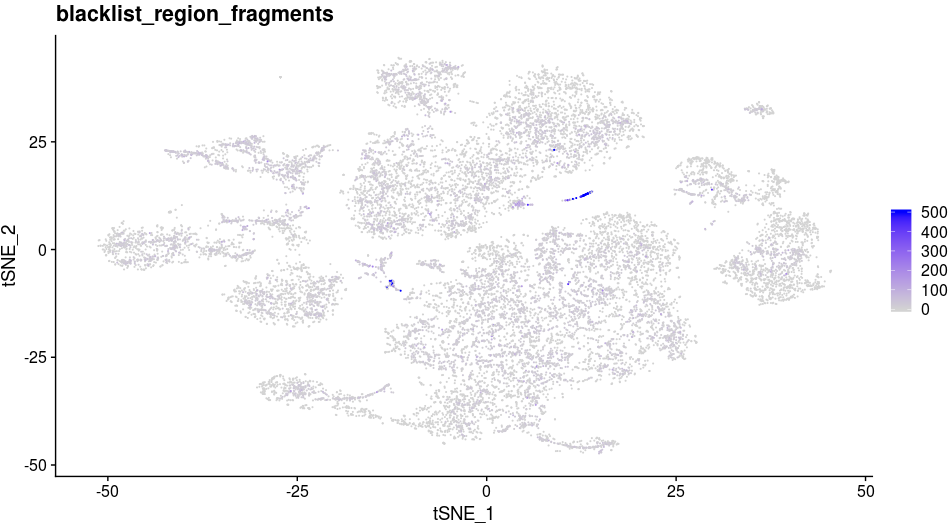
此外,你还可以发现B细胞前体类型附近有一类细胞只由scATAC-seq的细胞构成。通过展示这些细胞在CellRanger分析结果提供的黑名单位置所占read数,可以推测出这类细胞可能是死细胞,或者是其他scRNA-seq无法检测的人为因素。
1 | coembed$blacklist_region_fragments[is.na(coembed$blacklist_region_fragments)] <- 0 |
Seurat这种基于基因活跃度得分进行细胞类型预测,是否靠谱,开发者提供了如下几个证据
- 总体预测得分(
pbmc.atac$prediction.score.max)高,意味着用scRNA-seq定义细胞类型比较可靠 - 我们可以在scATC-seq降维结果中
- 利用相同锚点的贡嵌入分析,发现两类形态能很好的混合
- 将ATAC-seq数据根据聚类结果构建pseduo bulk, 发现和真实的bulk数据近似