NECAT是肖传乐老师团队开发的一个针对Nanopore数据组装的软件,目前该工具尚未发表,除了https://github.com/xiaochuanle/NECAT有软件的介绍外,暂时没有中文资料介绍NECAT的使用。
太长不看的结论: Nanopore的组装推荐用下NECAT。组装之后是先用MEDAKA做一遍三代polish,然后用NextPolish默认参数做二代polish。
这篇将会以一篇发表在Nature Communication上的拟南芥nanopore数据介绍如何使用NECAT进行组装,运行在CentOS Linux release 7.3.1611 (Core),64G为内存, 20线程(Intel(R) Xeon(R) CPU E5-2640 v4 @ 2.40GHz),下面是正文。
软件安装
NECAT可以在https://github.com/xiaochuanle/NECAT/releases/页面获取最新的软件下载地址,这里下载的是0.01版本。
1 | wget https://github.com/xiaochuanle/NECAT/releases/download/v0.01/necat_20190307_linux_amd64.tar.gz |
目前0.01版本不支持gz文件作为输入,但后续版本应该会支持。
目前更新到necat_20200119_Linux-amd64,新版本安装方法为
1 | wget https://github.com/xiaochuanle/NECAT/releases/download/SourceCodes20200119/necat_20200119_Linux-amd64.tar.gz |
新版本增加gz文件支持。目前测试发现文件名需要符合xxx.fastq或xxx.fastq.gz命名格式,对于fq.gz无法识别,会导致程序文件出错。
实战
第一步: 新建一个分析项目
1 | mkdir NECAT && cd NECAT |
以发表在NC上的拟南芥数据为例, 下载该数据
1 | # 三代测序 |
第二步: 创建配置文件
1 | necat.pl config ath_config.txt |
配置文件中,主要修改如下几个参数
1 | PROJECT=athaliana #项目名 |
参数中还有一个,NUM_ITER=2,它并非是简单的重复2次纠错,它的每一轮的校正目的其实不同,第一轮的优先级是敏感度(senstitive), 第二轮之后主要追求速度(fast)。
除了上面的配置参数外,其他参数可以不需要修改,使用默认的值即可。需要修改的话,参考最后的参数说明部分。
第三步: 序列纠错
1 | necat.pl correct ath_config.txt & |
纠错后的reads在athaliana/1-consensus/cns_final.fasta
cns_finla.fasta的统计信息会输出在屏幕中, 或者自己用fsa_rd_stat也能得到同样的结果
1 | Count: 206342 |
此外我还用time获取了运行时间,纠错花了大概一个小时。
1 | real 55m31.451s |
第四步: contig组装
1 | necat.pl assemble ath_config.txt & |
结果在athaliana/4-fsa/contigs.fasta
关于contigs.fata统计信息会输出在屏幕上,同样用fsa_rd_stat 也可以。
1 | Count: 162 |
时间用了75分钟
1 | real 74m53.127s |
第五步: contig搭桥
1 | necat.pl bridge ath_config.txt |
结果在athaliana/6-bridge_contigs/bridged_contigs.fasta
1 | Count: 127 |
从N50和N75可以看出这一步会提高组装的连续性。
组装结果polish
对Nanopore组装结果进行polish的常用软件有下面3个
由于拟南芥的基因组比较小,我分别用了Medaka和racon对输出结果进行polish(因为没有原始信号数据,因此nanopolish用不了),代码如下
Medaka
1 | NPROC=20 |
三轮Racon:
1 | gzip -dc ERR2173373.fastq.gz > ERR2173373.fastq |
在后续评估质量的时候,我发现单纯用三代polish的结果还不是很好,因此我用他们提供的二代测序,用NextPolish对NECAT的结果进行polish。
1 | # 二代测序 |
run.cfg内容如下, 其中sgs.fofn记录的就是解压后的ERR2173372_1.fastq和ERR2173372_2.fastq的路径
1 | [General] |
我考虑了两种情况,一种是直接用二代polish,另一种是三代polish之后接二代polish。
结果评估
在计算时间上,我之前用Canu跑了相同的数据,设置原始错误率0.5,纠错后错误率为0.144,用3个节点(每个节点12个线程),运行了3天时间,但是NECAT只需要3个小时左右就能完成相同的分析,这个速度差异实在是太明显了。
用Minimap2 + dotPlotly绘制CANU,NECAT和拟南芥参考基因组的共线性图
1 | minimap2 -t 20 -x asm5 Athaliana.fa NECAT.fa > NECAT.paf |
NECAT的结果

CANU的结果
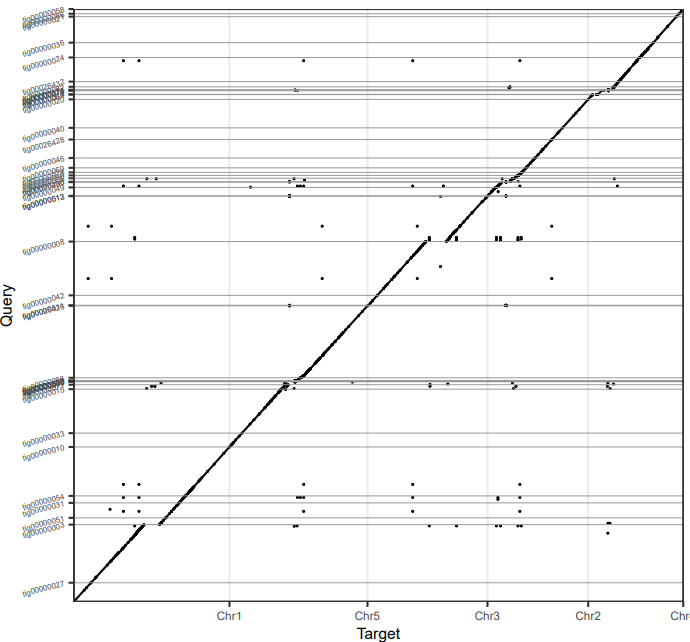
NECAT和CANU都和参考基因组有着良好的共线性,但是NECAT的连续性更好,几乎成一条直线。
之后,我使用了QUAST来评估Canu,NECAT初步组装,NECAT用Medaka, nanopolish和racon纠错的结果(MD: MEDAKA, RC: RACON, NP:NextPolish)。
1 | quast.py -t 100 --output-dir athaliana --circos \ |
一些描述基本信息
1 | CANU N50 = 4875070, L50 = 7, Total length = 114689024, GC % = 36.09 |
在BUSCO完整度上, 以embryophyta_odb10作为物种数据库, 其中ONTmin_IT4是发表的文章里的结果, Athalina则是拟南芥的参考基因组,我们以它们的BUSCO值作为参照。
1 | Athalina : C:98.6%[S:98.0%,D:0.6%],F:0.4%, M:1.0%, n:1375 |
二代Polish后的BUSCO结果如下(MD: MEDAKA, RC: RACON, NP:NextPolish):
1 | Athalina : C:98.6%[S:98.0%,D:0.6%],F:0.4%,M:1.0%,n:1375 |
从以上这些数据,你可以得到以下几个洞见:
- 在Nanopore的组装上,NECAT效果优于Canu,无论是连续性还是N50上
- MEDAKA三代polish效果好于RACON。在速度上,MEDAKA比三遍RACON都慢,并且MEDAKA会将一些可能的错误组装给打断
- Nanopore的数据用NECAT组装后似乎用NextPolish进行polish后就行,但是由于物种比较小,可能不具有代表性。
结论: Nanopore的组装建议用NECAT。组装之后是先用MEDAKA做一遍三代polish,然后用NextPolish默认参数做二代polish。
配置文件补充
这部分对配置文件做一点简单补充。
下面这些参数相对简单,不需要过多解释,按照自己需求修改
- CLEANUP: 运行完是否清理临时文件,默认是0,表示不清理
- USE_GRID: 是否使用多节点, 默认是false
- GRID_NODE: 使用多少个节点,默认是0,当USE_GRID为true时,按照自己实际情况设置
以下的参数则是需要根据到具体的软件中去查看具体含义,需要和软件开发者讨论
- OVLP_FAST_OPTIONS: 第二轮纠错时, 传给
oc2pmov - OVLP_SENSITIVE_OPTIONS: 第一轮纠错时, 传给
oc2pmov - CNS_FAST_OPTIONS: 第二轮纠错时,传给
oc2cns - CNS_SENSITIVE_OPTIONS: 第一轮纠错时,传给
oc2cns - TRIM_OVLP_OPTIONS: 传给
oc2asmpm - ASM_OVLP_OPTIONS: 传给
oc2asmpm - FSA_OL_FILTER_OPTIONS: 参数传给
fsa_ol_filter - FSA_ASSEMBLE_OPTIONS: 参数传给
fsa_assemble - FSA_CTG_BRIDGE_OPTIONS: 参数传给
fsa_ctg_bridge