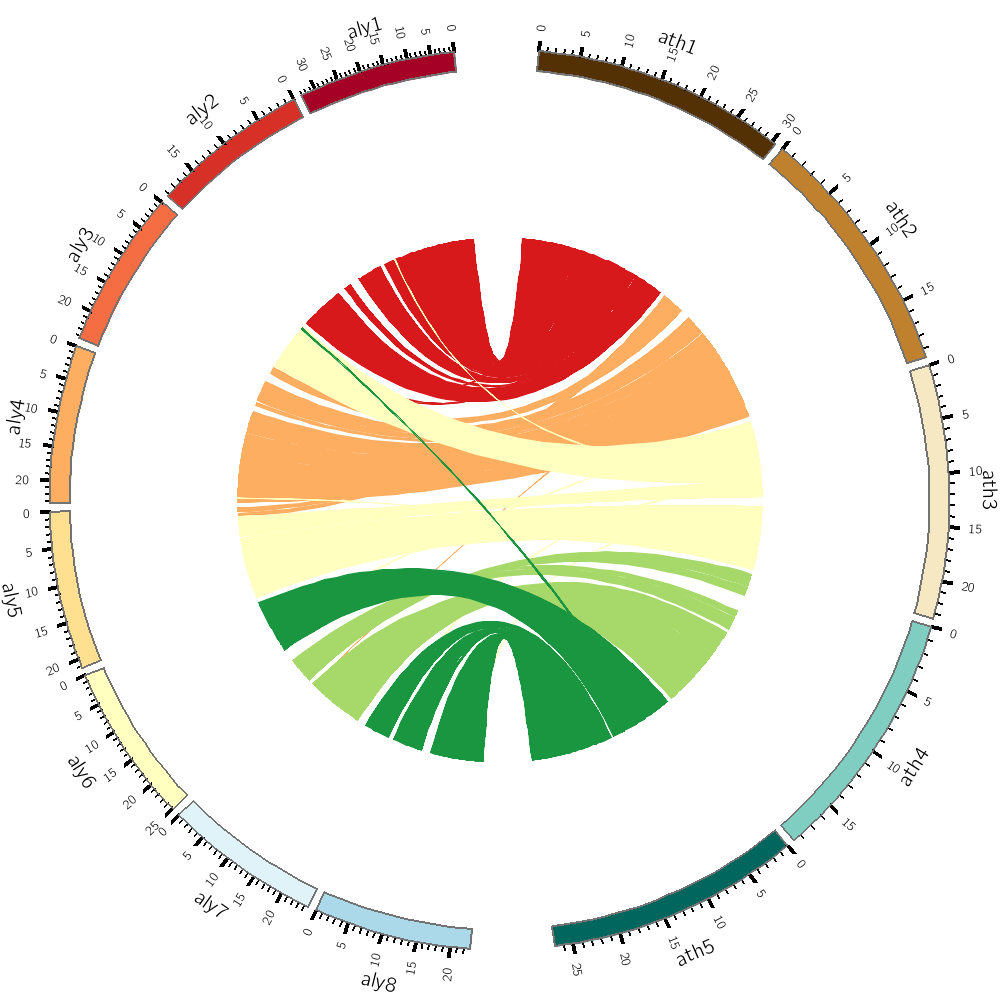
这一部分承接从零开始学CIRCOS绘制圈图(四),对之前的布局进行调整,使结构更加适合于发表。
染色体的位置顺序
默认情况下是会显示所有的染色体,而且会先从ath1到ath5,然后从aly1到aly8, 那么如何调整顺序呢?
需要设置的参数是chromosomes_order, 按照自己的需求调整位置。
1 | chromosomes_order = aly8,aly7,aly6,aly5,aly4,aly3,aly2,aly1 |
虽然circos提供了一些快捷操作,比如说-,$,^, 但是远不如直接输入直观。
反转染色体的坐标顺序
默认都是从0到最大值,可以通过正则匹配的方式,将aly的染色体改成从最大值到0
1 | chromosomes_reverse = /aly/ |
调整染色体缩放
为了让两个物种的基因组能够占Circos的两边,需要设置缩放。
1 | chromosomes_scale = /aly/=0.5rn,/ath/=0.5rn |
/aly/=0.5rn表示aly的所有染色体总共占据50%的空间
定义染色体的间隔
<spcaing>控制的是不同染色体之间间隔的空隙大小,比如说下面的参数表示所有染色体之间的间隔是0.005r。
1 | <spacing> |
我们设置某些染色体之间的距离大一些,比如说两个物种的起始染色体和两个物种的终止染色体
1 | <spacing> |
调整起始位置
默认情况下,ath1从-90度位置开始,由于设置了aly1和ath1之间的空隙为10u,于是看起来就不对称了。为了让结果图能够对错,需要调整角度。
1 | angle_offset* = -85 |
最终效果