在从零开始学CIRCOS绘制圈图(一), 我们已经绘制出一个比较丑的circos图,这一部分是讲解一些细节。
这一部分会从上一步的两个文件开始,分别是
karyotype.tair10.txt
1 | chr - chr1 chr1 0 30427617 chr1 |
circos.conf
1 | karyotype = karyotype.tair10.txt |
配色
第一个要解决的问题是配色问题,其实上一部分的配色定义依旧有点丑。如果不想重新打开文件修改的话,其实除了在karyotype文件里定义颜色外,我们还可以直接在circos.conf文件定义颜色.
举个例子,在karyotype = karyotype.tair10.txt后加一行
1 | chromosomes_color = chr1=rdylbu-11-div-1,chr2=rdylbu-11-div-3,chr3=rdylbu-11-div-5,chr4=rdylbu-11-div-7,chr5=rdylbu-11-div-9 |
问题是,我们怎们知道这些名字后所代表的颜色呢?
Circos中颜色的命名格式为PALETTE-NUMCOLORS-TYPE-IDX:
- PALETTE:调色版名,如rdylbu
- NUMCOLORS: 颜色数目, 11
- 调色版类型: div(diverging), seq(sequential), qual(qualitative)
- IDX: 调色版中的颜色索引
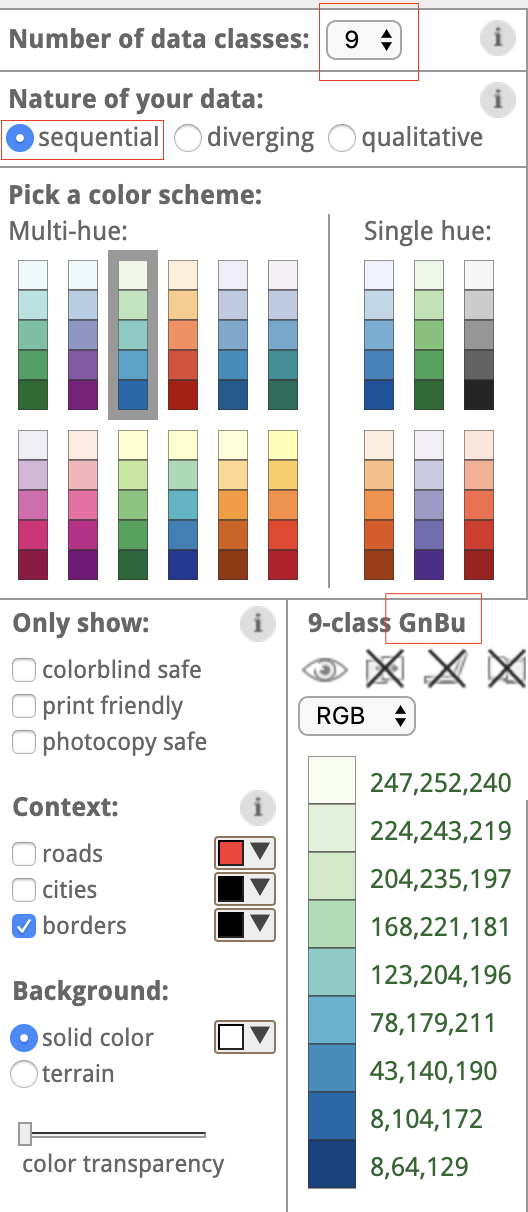
而Circos颜色来自于http://colorbrewer2.org
因此,gnbu-9-seq对应的是就是下图的9-class GnBu
配色不仅仅用在karyotype中,在后续的热图和柱状图等还会涉及到它,毕竟一张好看的图,配色占了很大的比例。
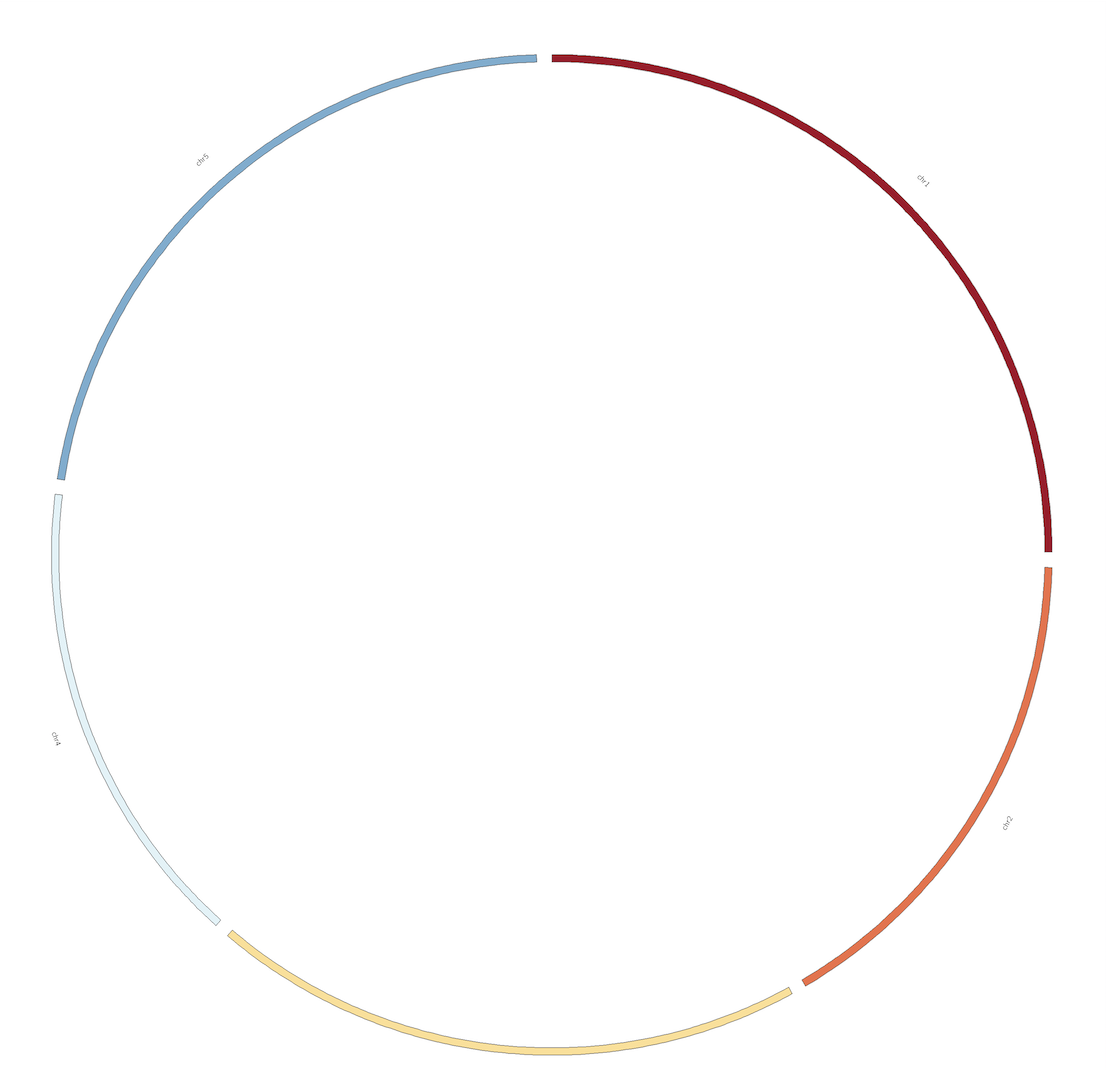
显示标签
默认输出图片是没有染色体名字的标签,需要在<ideogram>里添加和label有关的参数
1 | ... |
关于标签(label), 更详细的介绍在http://circos.ca/documentation/tutorials/ideograms/labels/
重新运行之后,发现字相对而言太小了。有两种结局方案,一种是调整label_size,比如说48,另一种是调整图片整体大小。
输出设置
默认情况下,输出图片的 半径是1500p, 所以设置的label_size=16就会显得特别小。我们可以在配置文件中调整图片的班级,以及其他设置。
1 | <image> |
注: 在参数后加一个*表示覆盖已有的设置,比如说svg*=no就是覆盖已有的svg=yes。
运行结果如下

刻度(ticks)
大部分的circos还会有刻度来展示染色体的大小。由于ticks的定制比较复杂,所以一般会单独搞一个配置文件,ticks.conf存放ticks相关的参数设置.
1 | chromosomes_units = 1000000 |
用 show_ticks和 show_tick_labels控制是否展示刻度,以及刻度对应的标签。
<ticks与</ticks>里控制刻度的全局参数,例如位置为1r(radius=1r), 颜色为黑色(color=black), 厚度为2p(thickness=2p), 由于默认直接展示染色体的实际位置,因此会显示1000000这种结果,所以定义multiplier=1e-6, 实际显示结果为 1000000 * 1e-6 = 1。
后面就可以通过<tick>和 </tick>来分别绘制不同类型的tick,重要的参数如下:
spacing表示刻度之间的距离,1u表示一个长度单位,需要在circos.conf文件里通过chromosome_unit来定义,通常都是chromosome_unit=1000000。size表示tick的长度show_label: 控制是否展示标签,默认不展示。label_offset: 则是让label往外在偏移一点距离
circos -conf circos.conf运行结果如下
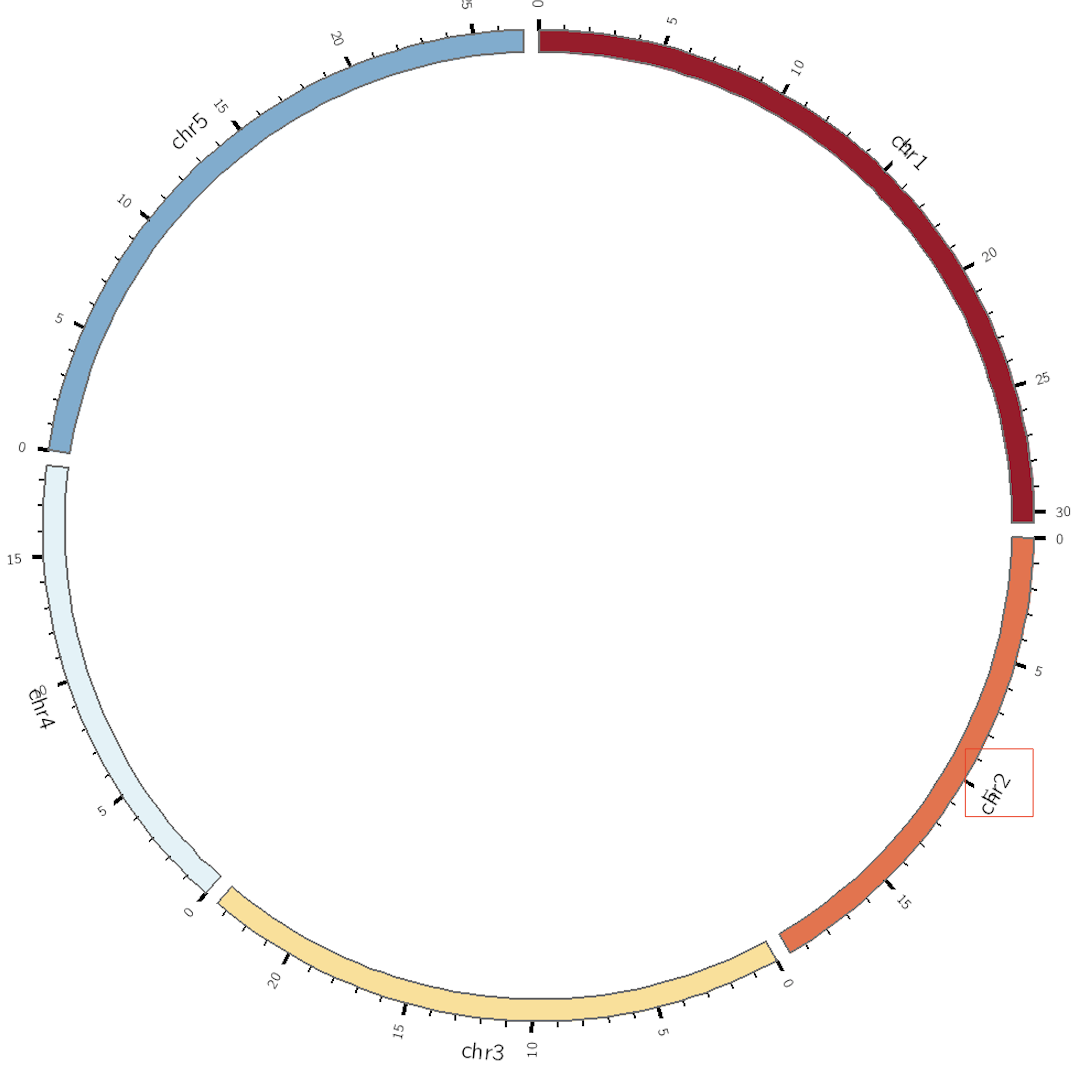
我们发现有些刻度的标签和染色体标签发生了重叠,这个可以通过label_radius进行调整。
单位
上面出现了控制图形不同元素大小的三个单位,p,r,u。p(pixels), 表示绝对大小, r(relative), 相对大小, u(chromosome unit)。 如果使用p作为单位,需要考虑最终输出图形<image>定义的radius。 而r是相对大小,会随着最终图形大小而发生变换。u一般在显示刻度时使用。
这一部分是在原来简陋的输出上进行了美化,没有用到除了染色体长度以外的信息。下一部分介绍如何展示基因密度等信息。