这篇文章是我发现canu输出的contig可能存在misassembly(错误组装), 为了探索这种错误是如何产生的,我尝试解决如下问题
- Canu输出contig的基本步骤
- contig和GFA是什么关系
- 如何提取contig对应的read
- 如何检查contig的graph
contig的构建过程
Canu的流程分为三个步骤,前两步是原始输入的纠错,最后一步是基于纠错后的reads来构建contigs。
步骤三的流程图
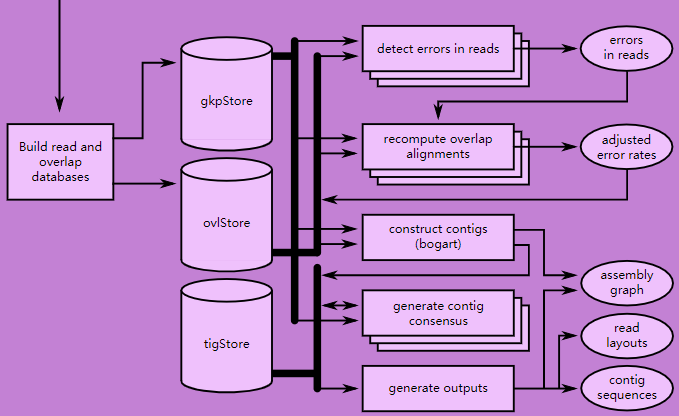
步骤三的输出文件
1 | 0-mercounts |
注: prefix表示文件名前缀。
根据流程和输出文件可知这一步骤可以细分为5步
- 构建read之间的overlap得到ovlStore
- 分析overlap检测错误
- 重新计算overlap的alignment
- 使用bogart构建contig
- 对contig进行consensus
- 构建输出, graph, layout, sequences
这里面最核心的是bogart, 它根据overlap情况构建graph,4-unitigger有如下的GFA文件,就是运行时产生的文件。
1 | prefix.best.edges.gfa |
bogart基于graph寻找最优路径, 最终得到unitig和contig两个结果,其中unitig更加碎一些,但更加准一些(Contigs, split at alternate paths in the graph)。
最终的contig还需要进一步的consensus,得到最终的输出
- prefix.contigs/unitigs.fasta: Fasta序列
- prefix.contigs/unitigs.gfa: contig.gfa已经在Canu1.9之后被移除,并非记录Fasta的构建过程
- prefix.contigs/unitigs.layout: 上述GFA并不存放序列,实际序列在layout中
- prefix.contigs/unitigs.layout.readToTig: contig ID和read ID的对应关系
- prefix.contigs/unitigs.layout.tigInfo: 每个contig的信息
- prefix.unitigs.bed: untig和contig的对应关系
Contig和GFA的关系
对于主目录输出结果中contig/untigs对应的fasta和gfa,我们需要明白其中fasta并非是由gfa生成,而是记录了contig与之前contig的overlap关系。
因此,如果用bandage可视化Canu 1.8之前的GFA文件,实际上你看到的是最终的contig之间的关系,而非read之间的关系。在Canu 1.9的更改日志中写道,
- Output file ‘contigs.gfa’ was removed because it was misleading
我们实际需要的是canu通过解析read之间的overlap关系图来得到最终的contig的gfa文件。而Canu并没有提供符合我们需求的文件,因为Canu输出的GFA文件中都没有以P为开头的记录,即没有记录contig的路径。
假如我对其中一个contig存在怀疑,那么我应该如何检查该contig对应read的overlap关系呢?
提取contig对应的read信息
注意,不同版本的Canu输出数据库未必兼容,也就是用和建立数据库不同版本的工具会报错。
以我自己的一个数据为例,对于.gfa中的一个编号,例如tig00000007,
1 | S tig00000007 * LN:i:62235 |
我们可知他的实际ID是7。它对应的read信息可以在.layout.readToTig找到。
1 | $ awk '$2==7' prefix.contigs.layout.readToTig | head -n 2 |
第一列即是contig对应的readID,例如632820
利用ovStoreDump我们就可以提取和此read overlap的所有read
1 | ovStoreDump -S prefix.seqStore -O unitigging/prefix.ovlStore -picture 632820 |
ovStoreDump可以以多种形式输出overlap的信息,例如-picture就是以ASCII展示overlap
或者根据readID用sqStoreDumpFASTQ提取实际的序列
1 | sqStoreDumpFASTQ -S prefix.seqStore -o - -fasta -r 632820 -raw | seqkit seq - | head |
-raw/-corrected/-trimmed表示提取不同阶段的结果
输出结果里使用的实际输入序列的ID编号,可以在prefix.seqStore/readNames.txt中找到readID和原始编号的对应关系。
掌握上述的操作,就可以提取指定的contig的read,然后将read回帖到该contig上,利用IGV可视化。
可视化检查contig的GFA
提取contig对应的readID, 然后根据readID抽取gfa
1 | contigID=$1 |
输出的.gfa就可以用Bandage进行可视化。但实际上发现这个操作并不太可行,不如直接上一节用IGV可视化alignment来的直接。