在「JCVI教程」使用JCVI进行基因组共线性分析(上)还是有一个尴尬的事情,就是只用到两个物种,不能展示出JCVI画图的方便之处,因此这里参考https://github.com/tanghaibao/jcvi/wiki/MCscan-(Python-version)的分析,只不过画图部分拓展下思路。
首先要运行如下代码获取目的数据
1 | python -m jcvi.apps.fetch phytozome Vvinifera,Ppersica,Tcacao |
然后按照教程的配置文件进行画图
layout文件内容如下
1 | # y, xstart, xend, rotation, color, label, va, bed |
seqids文件内容如下
1 | chr1,chr2,chr3,chr4,chr5,chr6,chr7,chr8,chr9,chr10,chr11,chr12,chr13,chr14,chr15,chr16,chr17,chr18,chr19 |
运行python -m jcvi.graphics.karyotype seqids layout会得到如下结果
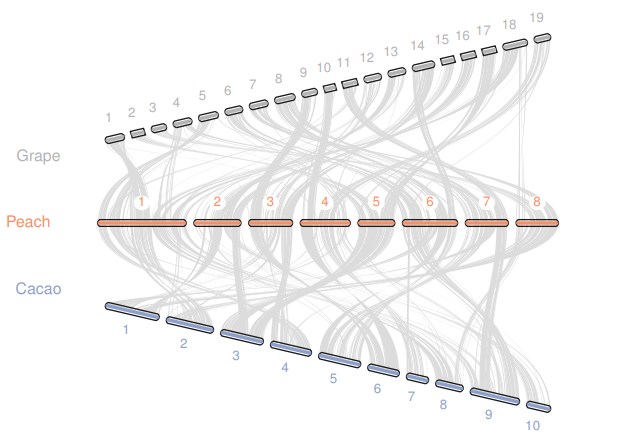
我就在思考一个问题如何让他形成一个三角形。经过一波三角运算和不断尝试,我定义了如下的layout
1 | # y, xstart, xend, rotation, color, label, va, bed |
那么效果怎么样呢?运行python -m jcvi.graphics.karyotype seqids layout吧
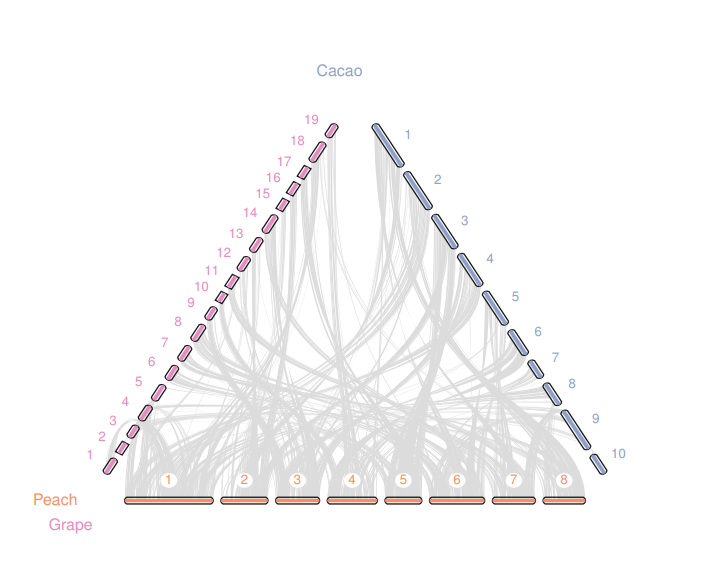
当然这里只展示了,grape和peach, peach和cacao之间的共线性,我又想着能不能加上grape和caocao呢?我尝试着运行下面的代码,
1 | python -m jcvi.compara.catalog ortholog grape cacao --cscore=.99 |
并继续修改了layout
1 | # y, xstart, xend, rotation, color, label, va, bed |
运行python -m jcvi.graphics.karyotype seqids layout会得到如下结果

我觉得给我一点时间,我也能用JCVI画出下面的图了