chromVAR是一个用于分析稀疏染色质开放的R包。chromVAR的输入文件包括,ATAC-seq处理后的fragments文件(过滤重复和低质量数据), DNAse-seq实验结果,以及基因组注释(例如motif位置)
chromVAR先根据所有细胞或者样本的平均情况来计算期望开放性, 然后用它来计算每个注释,每个细胞或样本的偏差,最后对开放进行纠正。
安装加载
安装R包
1 | BiocManager::install("GreenleafLab/chromVAR") |
chromVAR的运行需要先加载下面这些R包
1 | library(chromVAR) |
chromVAR能够使用多核进行并行运算,调用方法如下
1 | # 全部用户 |
注意: 不运行的话,后续代码可能会报错
读取输入
chromVAR接受的输入是落入开放区域的read数统计表。有许多软件可以做到,chromVAR也提供了相应的方法。
首先要提供一个peak文件,文档建议这个peak文件存放的peak为等宽非重叠,建议宽度在250-500 bp之间。peak文件可以利用已有的bulk ATAC-seq或DNAse-seq数据来获取。对于来自于多个样本的peak,需要先用filterPeaks函数保证peak之间不重合。
使用getPeaks读取peak
1 | peakfile <- system.file("extdata/test_bed.txt", package = "chromVAR") |
MACS2分析结果里会提供narrowpeak格式文件,chromVAR提供了readNarrowpeaks函数进行读取。
随后用getCounts函数基于BAM文件和加载的peak获取count
1 | bamfile <- system.file("extdata/test_RG.bam", package = "chromVAR") |
bamfile可以有多个bam文件路径,bam文件的类型和colData对应。此外by_rg定义是否要根据BAM文件中的RG对输入分组。
实际演示的时候,我们官方提供的示例数据
1 | data(example_counts, package = "chromVAR") |
因为这是一个*SummarizedExperiment 对象,因此我们就能用assay,colData和rowData 了解这个数据集
主体是一个存放count的dgCMatrix
1 | assay(example_counts)[1:5,1:2] |
行是特征(feature), 这里就是peak
1 | head(rowData(example_counts), n=2) |
列表示样本,
1 | head(colData(example_counts), n=2) |
前期准备
分析peak中的GC含量
GC含量信息用于确定哪些peak可能是背景, addGCBias返回更新后的SummarizedExperiment 。其中genome支持BSgenome, FaFile和DNAStringSet对象。
1 | library(BSgenome.Hsapiens.UCSC.hg19) |
过滤输入(单细胞)
如果是处理单细胞数据,建议过滤测序深度不够,或者是信噪比低不够(也就是peak中所占read比例低)的数据,主要靠两个参数,min_in_peaks和min_depth.
1 | counts_filtered <- filterSamples(example_counts, min_depth = 1500, |
然后我们用filterSamplesPlot对过滤情况进行可视化
1 | filtering_plot <- filterSamplesPlot(example_counts, min_depth = 1500, |

确认标准后,就可以过滤
1 | counts_filtered <- filterPeaks(counts_filtered, non_overlapping = TRUE) |
获取Motifs和分析包含motif的peak
可以用getJasparMotifs在JASPAR数据库中提取motif信息,但是其中getJasparMotifs只是TFBSTools::getMatrixSet一个简单的封装而已, 默认用的是JASPAR2016, 建议通过下面的代码获取最新版本的数据。
1 | # BiocManager::install("JASPAR2018") |
collection参数中接受: “CORE”, “CNE”, “PHYLOFACTS”, “SPLICE”, “POLII”, “FAM”, “PBM”, “PBM_HOMEO”, “PBM_HLH” 选项。
获取Motif的另外一种方式是用chromVARmotifs, 这个R包的主要功能就是整合了一些motif, 通过devtools::install_github("GreenleafLab/chromVARmotifs")进行安装。
之后用motifmatchr中的macthMotifs去分析peak中motif包含情况。默认会返回一个SummarizedExperiment 对象,就是一个稀疏矩阵,来提示是不是匹配了motif
1 | library(motifmatchr) |
关键参数是p.cutoff用于设置严格度,默认是0.00005其实能够返回比较合理的结果。如果需要一些额外信息,可以通过参数out来调整,例如out=positions表示返回实际的匹配位置
偏离度和变异度分析(Deviations and Variability )
上面都是准备阶段,计算deviations和Variability才是这个软件的重点。
计算偏离度
函数computeDeviations返回的SummarizedExperiment 对象, 包含两个”assays”,
- 偏差纠正后的开放偏离度:
assays(dev)$deviations - 偏离度的Z-score:
assays(deviations)$z: 考虑到了当从基因组上随机抽取一些GC含量类似的片段分析时,有多大概率会得到该结果。
1 | dev <- computeDeviations(object = counts_filtered, annotations = motif_ix) |
compuateDeviations支持背景peak的输入,用于的偏移度得分进行标准化,默认会自动计算不会返回结果。你可以选择用getBackgroundPeaks分析背景,然后传递给computeDeviations
1 | bg <- getBackgroundPeaks(object = counts_filtered) |
变异度
computeVariability会返回一个数据框data.frame,里面有变异度, 该变异度的bootstrap置信区间和衡量拒绝原假设的概率(即 > 1)
1 | variability <- computeVariability(dev) |
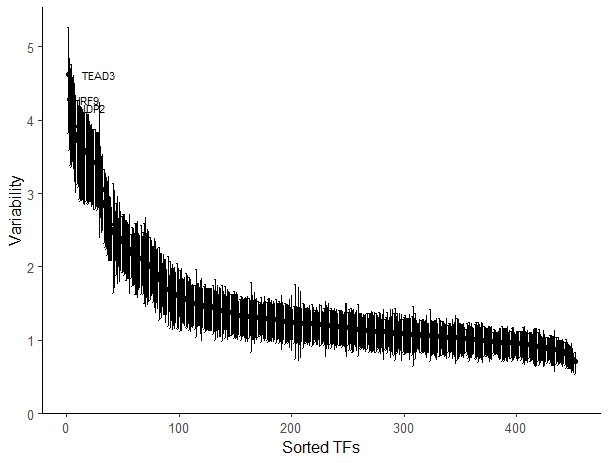
偏离度可视化
可以利用tSNE可视化偏离度
1 | tsne_results <- deviationsTsne(dev, threshold = 1.5, perplexity = 10) |

参考资料
- 数据库JASPAR是一个开放性数据库,用于存放人工审查的非冗余转录因子(TF)结合谱,数据格式为PFM(position frequency matrices ), TFFM( TF flexible models)
- chromVAR官方文档
- chromVAR: inferring transcription-factor-associated accessibility from single-cell epigenomic data